Nachwuchsforschungsgruppe | Medical Omics
Übersicht
Die Nachwuchsforschungsgruppe (NWG) Medical Omics entwickelt KI-basierte Tools für die Analyse großer biomedizinischer Datensätze aus Klinik und Grundlagenforschung. Um die Komplexität biologischer Systeme und ihrer physiologischen und pathologischen Zustände umfassend abzubilden, nutzen wir hierzu mehrere Datenmodalitäten, wie zum Beispiel Single-Cell RNA-Seq*, DNA Methylierung* und medizinische Bildgebung*. In enger Zusammenarbeit mit Kliniker.innen an der Charité entwickeln wir Systeme, die Ärzt:innen bei der Diagnose und Behandlungsplanung von Tumorerkrankungen unterstützen und Routineaufgaben, wie die Annotation* radiologischer Bildgebung, übernehmen können. Um eine bessere Anbindung an die in den Kliniken vorhandene Expertise zu gewährleisten, arbeiten wir außerdem an Feedbacksystemen, die gezielt das Training unserer KI-Modelle verbessern können.
Begriffserklärungen:
*Single-Cell RNA-Seq:
Mit diesem Verfahren wird parallel die Expression (vereinfacht: Aktivität) aller Gene in einer Zelle gemessen. Moderne Verfahren können dies parallel für tausende Zellen gleichzeitig durchführen, um beispielsweise die Interaktion von Immunzellen mit infiziertem Gewebe erforschen zu können.
*DNA Methylierung:
Jede Zelle eines Organismus enthält die gleiche Genominformation. Unterschiedliche Zelltypen im Körper entstehen also aus der verschiedenen Aktivierung (Expression) derselben Gene. Die DNA inaktiver Gene ist häufig durch Methylierung chemisch verändert. Aus der Messung dieser Methylierung kann die zelluläre Herkunft eines Stücks DNA ermittelt werden.
*Medizinische Bildgebung:
Dieser Begriff umfasst eine Vielzahl von Verfahren, unter anderem Röntgenaufnahmen, Computertomographie (CT) und Magnetresonanztomo-graphie (MRT).
*Annotation:
In einem Bild werden bestimmte Strukturen händisch eingezeichnet.
*Tiefe neuronale Netze:
Hierbei handelt es sich um ein Verfahren des maschinellen Lernens, bei dem ein aus mehreren Ebenen bestehendes mathematisches Modell erlernt, auf Basis von Daten Vorhersagen zu treffen. Die Verschaltung der einzelnen Ebenen des Modells hat gewisse Ähnlichkeit mit der Kommunikation einzelner Nervenzellen des Gehirns.
*Nanopore-Sequenzierung:
Bei der Nanopore-Sequenzierung wird die Sequenz eines Stücks DNA ermittelt, indem dieses durch die Poren einer Membran gezogen wird, an der eine Spannung anliegt. Da die einzelnen Basen („Buchstaben“) der DNA unterschiedlich groß sind, verändert sich je nachdem, welche Base sich gerade in der Pore befindet, deren elektrischer Widerstand. Aus dieser Veränderung des Widerstands lässt sich sowohl die Sequenz der DNA als auch das Vorhandensein eventueller chemischer Modifikationen ablesen.
*Genexpressionsprofile:
Die Aktivität eines oder mehrerer Gene in allen Zellen einer Probe.

Themenfelder
Einzelzell-Sequenzierung
Gen-Set-Inferenz
Im Gegensatz zu klassischen sogenannten „bulk“ Sequenziermethoden, die gemittelte Genexpressionsprofile* liefern, kann Einzelzell-Sequenzierung die Genexpression innerhalb eines Gewebes mit bislang ungekannter Genauigkeit auflösen. Die Menge an oft stark rauschenden und unvollständigen Daten verursacht jedoch bei der Analyse größeren Aufwand und erfordert neue, maßgeschneiderte Auswertungsverfahren. Wir haben ein Deep-Learning-basiertes Tool entwickelt, das Muster und zusammengehörige Sets von Genen in Single-Cell RNA-Seq* Daten entdeckt und diese Sets biologischen Zuständen und Zelltypen zuordnen kann. Dies ermöglicht es Wissenschaftler.innen, schnell eine Intuition bezüglich der in einem Gewebe ablaufenden Prozesse zu entwickeln.
COVID-19
Während der COVID-19 Pandemie haben wir gemeinsam mit anderen Gruppen an der Charité - Universitätsmedizin Berlin, dem Universitätsklinikum Leipzig und dem Deutschen Krebsforschungszentrum (DKFZ) einige der ersten Einzelzellreferenzatlanten von gesundem und von SARS-CoV-2 befallenem Lungengewebe erstellt. Wir konnten zeigen, weshalb einzelne Gruppen ein höheres Risiko haben, schwer an COVID-19 zu erkranken, während bei Kindern im Allgemeinen ein abgeschwächter Verlauf zu beobachten ist. Über rein wissenschaftliche Erkenntnisse hinaus haben diese Studien in einer klinischen Phase II Studie gemündet, die ein potenzielles Medikament für die Behandlung von schweren COVID-19-Krankheitsverläufen erprobt.
Andere Bereiche
Abgesehen von diesen zwei Bereichen ist die NWG in eine größere Zahl von Projekten involviert, die Themenfelder von Herzrhythmusstörungen bis hin zu Spermatogenese (Bildung von Samenzellen aus Keimzellen) abdecken. Darüber hinaus findet eine aktive Entwicklung neuer Methoden für die Analyse von Einzelzellsequenzierdaten statt.
Glioblastome
Mehrere aktuelle Projekte beschäftigen sich mit Glioblastomen, den häufigsten und aggressivsten der malignen primären Hirntumore in Erwachsenen.
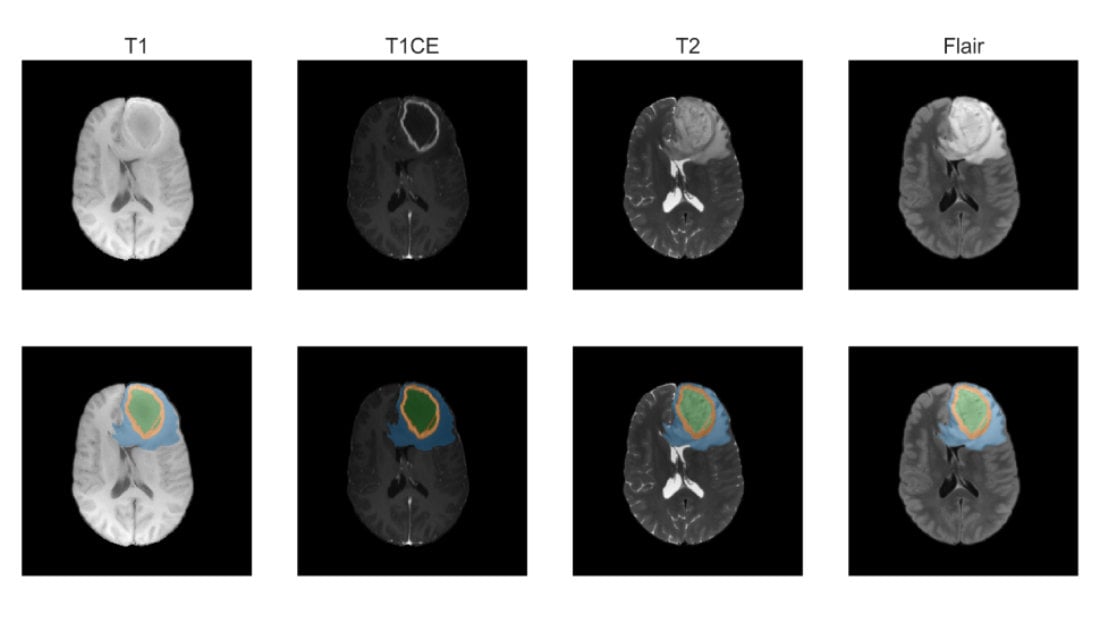
Medizinische Bildgebung
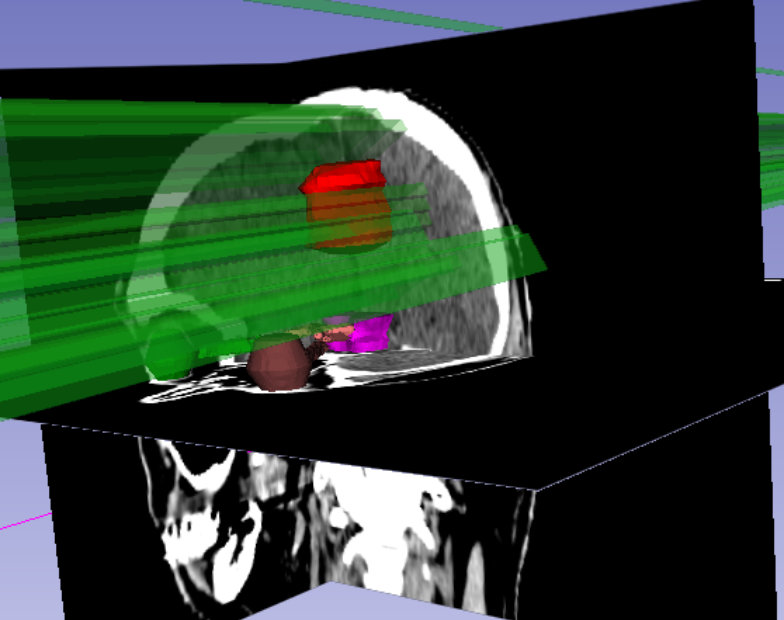
Um operative Entfernung und Bestrahlung von Tumoren zu planen, muss die genaue Position der durch den Eingriff gefährdeten Organe (organs at risk, OARs) sowie des Tumors mittels 3-dimensionaler CT oder MRT Bildgebung bestimmt werden. Diese Positionen manuell zu annotieren*, ist zeit- und arbeitsaufwändig. Um diesen Prozess zu automatisieren, haben wir tiefe neuronale Netze* entwickelt, welche OARs und Hirntumore innerhalb von Sekunden mit höchster Genauigkeit segmentieren können.
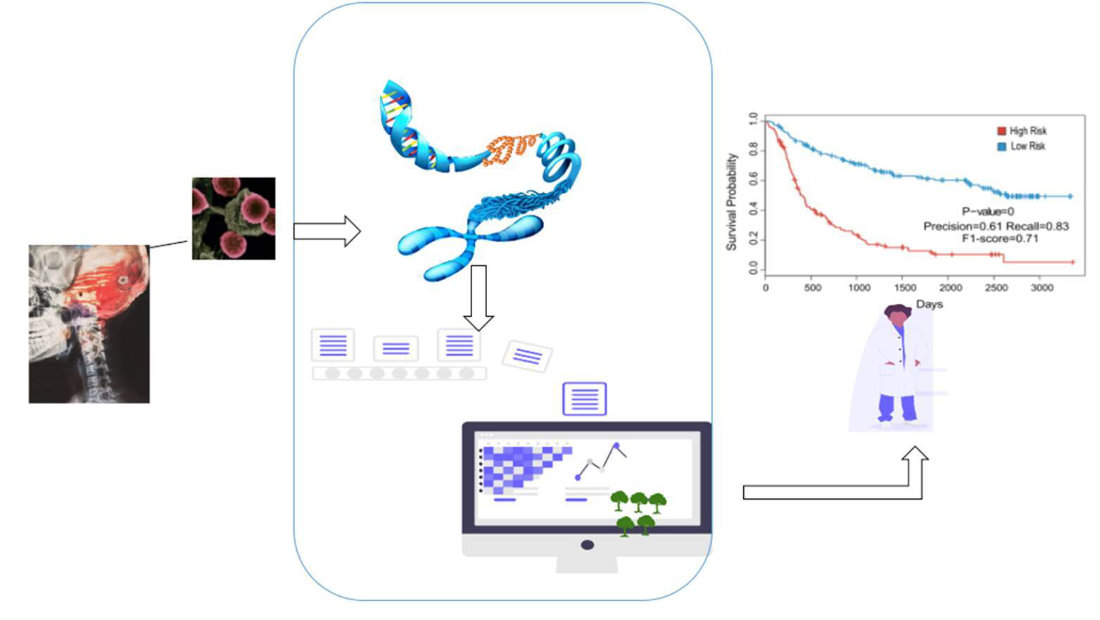
Darüber hinaus arbeiten wir an der Erstellung von Modellen, welche die Überlebenszeit eines Patienten in Abhängigkeit von der gewählten Therapieoption vorhersagen können und somit eine für den einzelnen Patienten maßgeschneiderte Therapie ermöglichen, die Erfolg und Risiko bestmöglich balanciert. Hierzu werden verschiedene Datenquellen, beispielsweise klinische Bildgebung*, DNA-Methylierung* und weitere Metadaten verwendet.
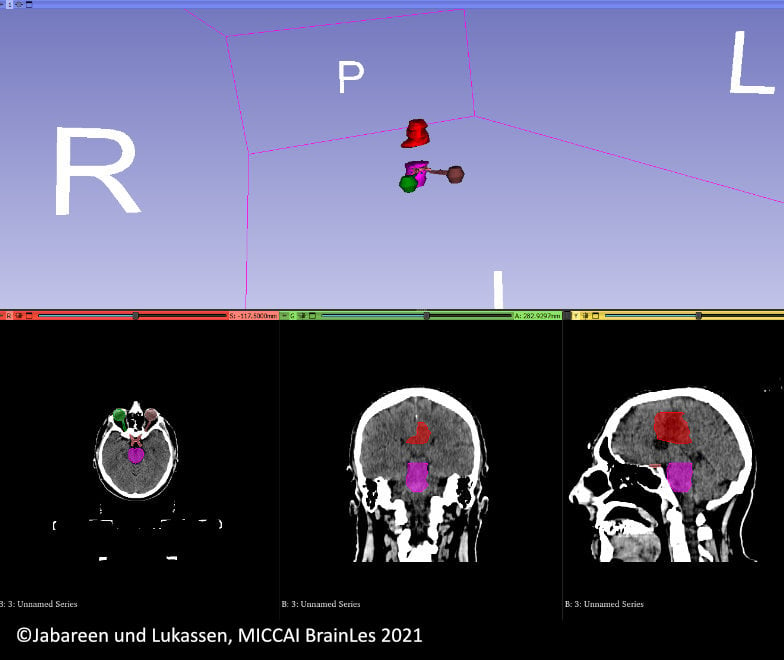
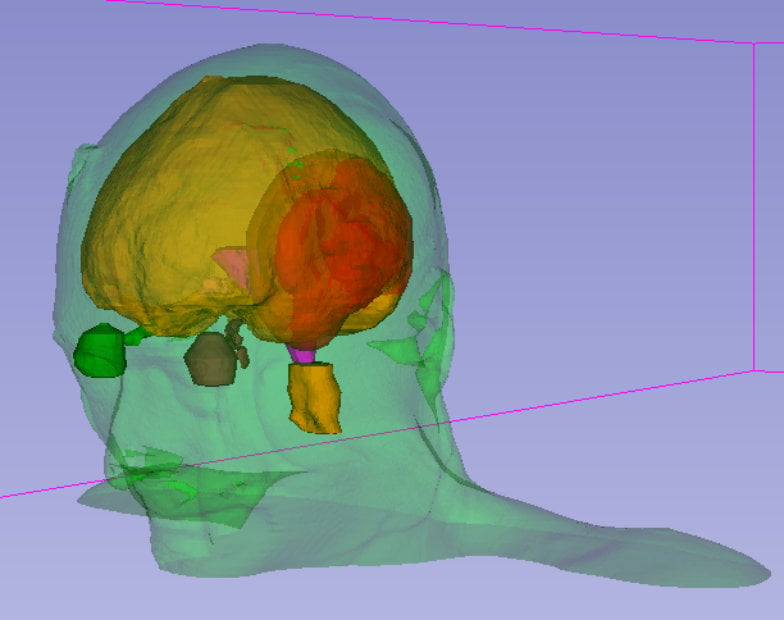
DNA-Methylierung
DNA-Methylierung* spielt in der Pathogenese (Entstehung und Entwicklung einer Krankheit) von Glioblastomen eine essenzielle Rolle. Beispielsweise kann sie die Mengen von DNA-Reparaturenzymen modulieren und dadurch über den Erfolg einer Therapie entscheiden. Darüber hinaus können verschiedene Subtypen von Glioblastomen anhand ihres DNA-Methylierungsstatus voneinander unterschieden werden.
Durch Verbesserungen in den letzten Jahren erreicht Nanopore Sequenzierung mittlerweile ein ausreichendes Qualitätsniveau, um verlässliche, wenn auch bruchstückhafte Informationen über den Methylierungs-status eines Tumors zu gewinnen. Um den Einsatz dieser überaus schnellen und relativ günstigen Technik in einer klinischen Umgebung zu ermöglichen, arbeiten wir gemeinsam mit Kooperationspartnern an der Charité an der Entwicklung von darauf optimierten Methoden des maschinellen Lernens.
Multi-omics
Das übergeordnete Ziel ist es, die einzelnen Unterprojekte zum Glioblastom zu einem Framework zu vereinigen, das die einzelnen Stufen der Diagnose und Behandlung von Patient:innen mit dieser Erkrankung abdeckt und dabei durch die Integration multipler Datentypen einen bestmöglichen Outcome für die Patienten ermöglicht.
Leiter der Nachwuchsforschungsgruppe Medical Omics
Dr. Sören Lukassen
Digital Health Center | Berlin Institute of Health (BIH) an der Charité – Universitätsmedizin Berlin
Mitglieder der NWG Medical Omics
Nabil Jabareen
Doktorand | Charité - Universitätsmedizin Berlin
Dongsheng Yuan
Doktorand | Charité – Universitätsmedizin Berlin


